Vorhersage des Verhaltens eines Proteins aus seinem Aussehen heraus
Proteine sind die Bausteine des Lebens und spielen eine Schlüsselrolle bei allen biologischen Prozessen. Das Verständnis, wie sie mit ihrer Umwelt interagieren, ist daher entscheidend für die Entwicklung effektiver Therapeutika und die Grundlage für die Gestaltung künstlicher Zellen.
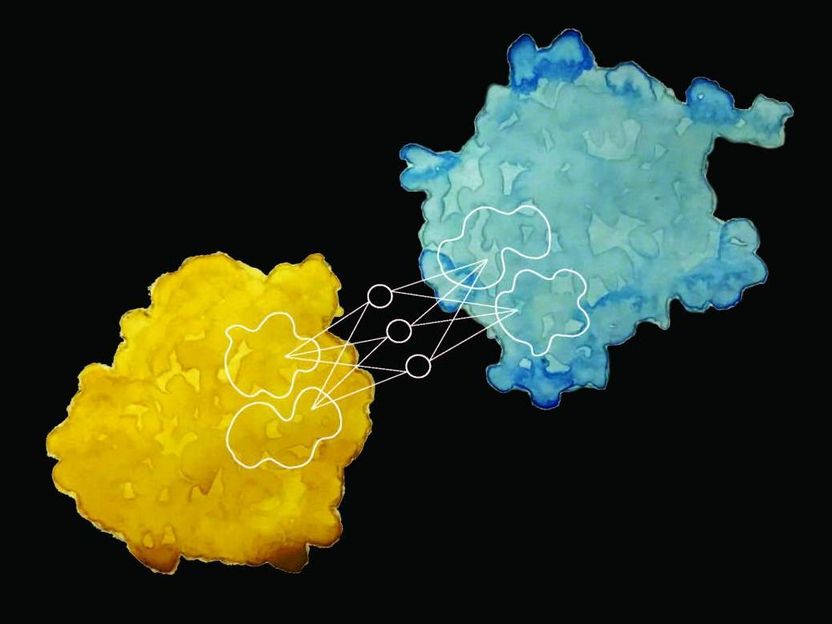
Forscher der EPFL haben eine neue Methode entwickelt, um die Wechselwirkungen eines Proteins mit anderen Proteinen und Biomolekülen sowie seine biochemische Aktivität allein durch die Beobachtung seiner Oberfläche vorherzusagen.
Laura Persat / 2019 EPFL
Forscher des Laboratory of Protein Design & Immunoengineering (LPDI), Teil des EPFL's Institute of Bioengineering at the School of Engineering, haben in Zusammenarbeit mit Mitarbeitern von USI-Lugano, Imperial College und der Twitter's Graph Learning Research Abteilung eine bahnbrechende maschinenlerngetriebene Technik entwickelt, um diese Wechselwirkungen vorherzusagen und die biochemische Aktivität eines Proteins allein anhand der Oberflächenbeschaffenheit zu beschreiben. Neben der Vertiefung des Verständnisses der Funktionsweise von Proteinen könnte die als MaSIF bekannte Methode auch die Entwicklung von proteinbasierten Komponenten für die künstlichen Zellen von morgen unterstützen.
Datengetriebene Forschung
Die Forscher nahmen einen riesigen Satz von Proteinoberflächendaten und fütterten die chemischen und geometrischen Eigenschaften in einen maschinell lernenden Algorithmus und trainierten ihn, diese Eigenschaften mit bestimmten Verhaltensmustern und biochemischer Aktivität abzugleichen. Anschließend nutzten sie die restlichen Daten, um den Algorithmus zu testen. "Durch das Scannen der Oberfläche eines Proteins kann unser Verfahren einen Fingerabdruck definieren, der dann zwischen den Proteinen verglichen werden kann", sagt Pablo Gainza, der erste Autor der Studie.
Das Team fand heraus, dass Proteine, die ähnliche Interaktionen durchführen, gemeinsame "Fingerabdrücke" haben.
"Der Algorithmus kann Milliarden von Proteinoberflächen pro Sekunde analysieren", sagt LPDI-Direktor Bruno Correia. "Unsere Forschung hat signifikante Auswirkungen auf das künstliche Proteindesign, so dass wir ein Protein so programmieren können, dass es sich auf eine bestimmte Weise verhält, indem wir lediglich seine oberflächenchemischen und geometrischen Eigenschaften verändern."
Das im Open-Source-Format veröffentlichte Verfahren könnte auch zur Analyse der Oberflächenstruktur anderer Molekültypen eingesetzt werden.
Hinweis: Dieser Artikel wurde mit einem Computersystem ohne menschlichen Eingriff übersetzt. LUMITOS bietet diese automatischen Übersetzungen an, um eine größere Bandbreite an aktuellen Nachrichten zu präsentieren. Da dieser Artikel mit automatischer Übersetzung übersetzt wurde, ist es möglich, dass er Fehler im Vokabular, in der Syntax oder in der Grammatik enthält. Den ursprünglichen Artikel in Englisch finden Sie hier.
Originalveröffentlichung
P. Gainza, F. Sverrisson, F. Monti, E. Rodolà, D. Boscaini, M. M. Bronstein, and B. E. Correia; "Deciphering interaction fingerprints from protein molecular surfaces using geometric deep learning"; Nature Methods; 2019.
Verwandte Inhalte finden Sie in den Themenwelten
Themenwelt Proteinanalytik
Die Proteinanalytik ermöglicht einen tiefen Einblick in diese komplexen Makromoleküle, ihre Struktur, Funktion und Wechselwirkungen. Sie ist unerlässlich für die Entdeckung und Entwicklung von Biopharmazeutika, das Verständnis von Krankheitsmechanismen und die Identifizierung von therapeutischen Zielen. Durch Techniken wie Massenspektrometrie, Western Blot und Immunoassays können Forscher Proteine auf molekularer Ebene charakterisieren, ihre Konzentration bestimmen und mögliche Modifikationen identifizieren.

Themenwelt Proteinanalytik
Die Proteinanalytik ermöglicht einen tiefen Einblick in diese komplexen Makromoleküle, ihre Struktur, Funktion und Wechselwirkungen. Sie ist unerlässlich für die Entdeckung und Entwicklung von Biopharmazeutika, das Verständnis von Krankheitsmechanismen und die Identifizierung von therapeutischen Zielen. Durch Techniken wie Massenspektrometrie, Western Blot und Immunoassays können Forscher Proteine auf molekularer Ebene charakterisieren, ihre Konzentration bestimmen und mögliche Modifikationen identifizieren.