Struktur von Phagen-DNA-Tunnel in atomarer Auflösung aufgeklärt
Methodischer Meilenstein gelungen
Phagen können Bakterien vernichten und sind darum für die Wissenschaft von großem Interesse. Grundlagenforscher vom Leibniz-Forschungsinstitut für Molekulare Pharmakologie (FMP) in Berlin interessieren sich insbesondere für den Tunnel, durch den die Phagen ihre DNA in Bakterien einschleusen. Nun haben sie zusammen mit Kollegen des Forschungszentrums Jülich und des Universitätsklinikums Jena die 3D-Struktur dieser entscheidenden Phagen-Komponente in atomarer Auflösung aufgeklärt. Die Kombination zweier Methoden – der Festkörper-NMR und der Kryoelektronenmikroskopie – war der Schlüssel zum Erfolg. Die Arbeit ist soeben im Fachmagazin „Nature Communications“ erschienen.
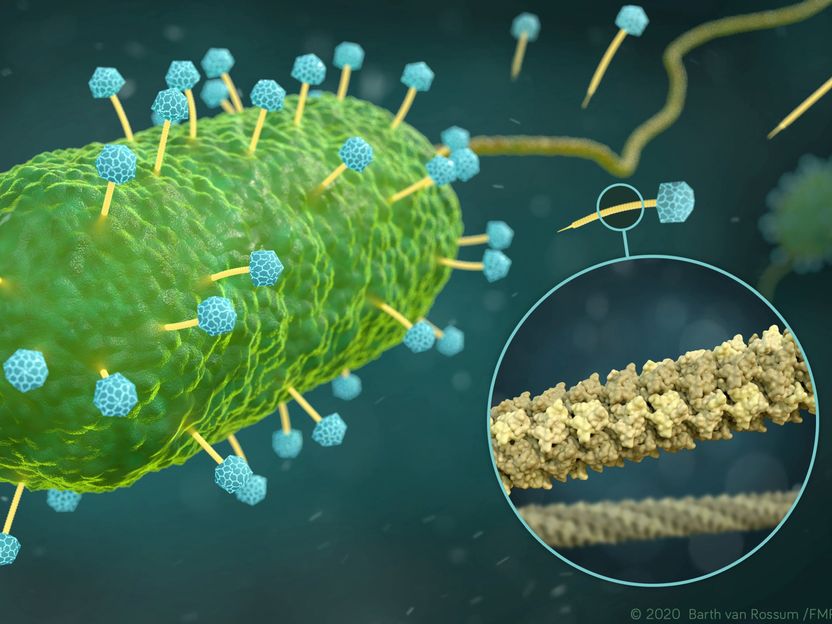
Künstlerische Darstellung von Phagen der Familie Siphoviridae (gelb und blau), die ein Bakterium (grün) infizieren. Der vergrößerte Bildausschnitt (Kreis) zeigt die atomare Struktur des DNA Tunnels (gelb), durch welchen die Phagen DNA in das Bakterium injiziert wird.
Visualisierung: Barth van Rossum, FMP
Im Zuge der zunehmenden Antibiotikaresistenzen sind Phagen verstärkt in den Blick der Forschung geraten. Phagen sind natürlich vorkommende Viren, die eine sehr nützliche Eigenschaft besitzen: Sie schleusen ihre DNA in Bakterien ein und vermehren sich dort, bis die bakterielle Zelle schließlich vernichtet wird. Deswegen spricht man auch von Bakteriophagen (dt. „Bakterienfresser“).
Dass sich mit diesem Ansatz multiresistente Keime bekämpfen lassen, wurde bereits gezeigt. Schlagzeilen machte im vergangenen Jahr der Fall eines Mädchens aus England, das mit genetisch modifizierten Phagen von einer schweren, mit Antibiotika nicht mehr therapierbaren Infektion geheilt werden konnte.
Doch von einer breiten Anwendung ist die Phagentherapie noch weit entfernt. Viele Grundlagen sind noch nicht verstanden, die wichtig für die weitere Therapieentwicklung wären. So war bislang unklar, wie die genaue Architektur jenes Tunnels aussieht, über den die Phagen ihre DNA in die Bakterien einschleusen. Nun haben Wissenschaftler vom Leibniz-Forschungsinstitut für Molekulare Pharmakologie (FMP) in Berlin gemeinsam mit Kollegen des Forschungszentrums Jülich und des Universitätsklinikums Jena die 3D-Struktur dieser entscheidenden Phagen-Komponente in atomarer Auflösung aufklären können.
Designt für den Transport von DNA
„Der DNA-Tunnel, der sich an das ikosaeder-förmige Kapsid anschließt, erinnert in seinem Aufbau und seiner Flexibilität ein wenig an eine Wirbelsäule“, beschreibt Prof. Adam Lange vom FMP, eine der neuen Einsichten. „Er scheint perfekt für den Transport von DNA designt zu sein.“
Die faszinierenden Einblicke in die Struktur und Operationsweise dieses raffinierten DNA-Transportwegs – in diesem Fall von einer Variante des Phagens SPP1 – konnten die Forscher gewinnen, weil sie auf eine innovative Weise die Festkörper-NMR und die Kryoelektronenmikroskopie (Kryo-EM) miteinander kombinierten. Die Kernspinresonanzspektroskopie (NMR) wurde von Langes Arbeitsgruppe speziell für diese Aufgabe im Rahmen eines ERC-Grants weiterentwickelt; die elektronenmikroskopischen Untersuchungen führte Kryo-EM-Experte Prof. Gunnar Schröder vom Forschungszentrum Jülich durch. Zusätzlich waren für die computergestützte Kombination beider Datensätze zur Strukturbestimmung neue Modellierungsalgorithmen notwendig, die von Prof. Michael Habeck vom Universitätsklinikum Jena entwickelt wurden. „Die Kombination der beiden Methoden war der Schlüssel zum Erfolg und ist ein methodischer Meilenstein“, betont Prof. Lange.
Während sich mit der Festkörper-NMR vor allem flexible Strukturen und winzigste Details gut darstellen lassen, erlaubt die Kryo-EM den Überblick über die Gesamtarchitektur. Das sich daraus ergebende Bild zeigt, dass sich sechs gp17.1-Proteine ringförmig organisieren, diese Ringe übereinander sitzen und so einen hohlen Tunnel formen. Hierbei sind die Ringe über flexible Linker miteinander verbunden, wodurch der Tunnel sehr biegsam wird. „Wir können nun verstehen, wie sich die negativ geladene DNA an der ebenfalls negativ geladenen Innenwand des flexiblen Tunnels abstößt und da reibungslos durchgleitet“, erläutert Maximilian Zinke vom FMP, Erstautor der jetzt in Nature Communications erschienen Studie. „Über diesen Weg werden die Bakterien letztendlich zerstört.“
Meilenstein für die integrierte Strukturbiologie
Nach Ansicht von Arbeitsgruppenleiter Adam Lange hat die Arbeit nicht nur die Phagenforschung einen guten Schritt vorangebracht, sondern auch die „integrierte Strukturbiologie“, wie die Kombination der beiden sich ergänzenden Methoden bezeichnet wird.
Dank des neuen Hochleistungs-Elektronenmikroskops Titan/Krios befindet sich die dafür nötige Infrastruktur seit kurzem auch auf dem Campus Berlin-Buch. Zudem sollen die schon vorhandenen NMR-Spektrometer demnächst um ein 1,2 Gigahertz-Gerät ergänzt werden. „Mit der Kryo-EM und dem weltweit leistungsstärksten NMR-Spektrometer werden wir künftig in der integrativen Strukturbiologie sehr präsent sein“, freut sich Adam Lange. „Das sind tolle Aussichten für den Campus und für den Forschungsstandort Berlin.“
Originalveröffentlichung
M. Zinke, K. A. Sachowsky, C. Öster, S. Zinn-Justin, R. Ravelli, G. F. Schröder, M. Habeck, A. Lange; "Architecture of the flexible tail tube of bacteriophage SPP1"; Nature Communications