Virtuelles Screening entdeckt Naturstoffe, die die Hauptprotease des SARS-CoV-2-Virus hemmen
Kampf gegen die COVID-19-Pandemie
Der Kampf gegen die COVID-19-Pandemie ist eine internationale wissenschaftliche Herausforderung, um Strategien zur Bekämpfung der Virusinfektion und zur Verhinderung ihrer Folgen zu finden. Neben den von den Gesundheitsbehörden zugelassenen Impfstoffen und Arzneimitteln ist es von entscheidender Bedeutung, neue Behandlungsmethoden zur Bekämpfung von Virusinfektionen zu finden, wenn diese bereits aufgetreten sind.
Dieser neue wissenschaftliche Fortschritt basiert auf dem virtuellen (in silico) Screening, einer computergestützten Analysetechnik zur Auswahl von Kandidatenmolekülen für künftige Arzneimittelkandidaten in einer chemischen Bibliothek.
Universidad de Barcelona
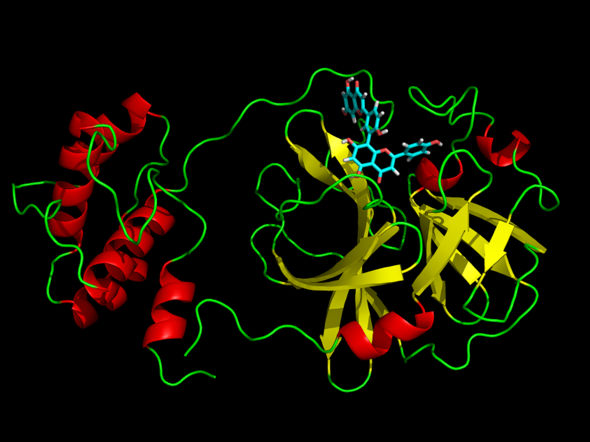
Eine im Journal of Chemical Information and Modeling veröffentlichte Studie hat eine Reihe von Naturstoffen identifiziert, die in der Lage sind, Mpro, die Hauptprotease des SARS-CoV-2-Virus, zu hemmen. Mpro ist ein Nicht-Strukturprotein, das eine wesentliche Rolle bei der Replikation und Transkription dieses Virus spielt und als potenzielles therapeutisches Ziel angesehen wird, da seine Hemmung das Fortschreiten des Virus verhindern könnte.
Die Arbeit steht unter der Leitung von Professor Jaime Rubio Martínez von der Fakultät für Chemie und dem Institut für theoretische und computergestützte Chemie (IQTC) der Universität Barcelona und könnte neue Perspektiven für die Entwicklung neuer therapeutischer Strategien im Kampf gegen COVID-19 eröffnen. Dieser neue wissenschaftliche Fortschritt basiert auf dem Einsatz von virtuellem Screening(in silico), einer Technik zur computergestützten Analyse von Molekülen, die in Datenbanken gespeichert sind, um potenziell pharmakologisch interessante Kandidaten zu identifizieren und auszuwählen.
Virtuelles Screening: auf der Suche nach zukünftigen Medikamenten
Heute besteht der klinische Ansatz bei COVID-19 in der symptomatischen Behandlung mit entzündungshemmenden Mitteln (z. B. Dexamethason oder Zytokininhibitoren) in Kombination mit Antibiotika zur Behandlung von Sekundärinfektionen. In jüngster Zeit wurde auch das Virostatikum Ritonavir zugelassen, aber es besteht weiterhin Bedarf an neuen Medikamenten. Im Zusammenhang mit der globalen Gesundheit besteht ein dringender Bedarf an der Entwicklung gezielter antiviraler Therapien gegen SARS-CoV-2, was zur Einführung verschiedener Strategien zur Identifizierung bioaktiver Verbindungen geführt hat, die als therapeutische Wirkstoffe eingesetzt werden können (einschließlich verfügbarer Medikamente und Naturprodukte). In diesem Zusammenhang ist das virtuelle Screening ein zuverlässiges, schnelles und effizientes Verfahren zur Entdeckung bioaktiver Verbindungen aus großen chemischen Sammlungen gegen ein bestimmtes molekulares Ziel.
Im Rahmen dieser Arbeit hat das Team ein virtuelles Screening der Selleck-Naturstoffdatenbank - einer chemischen Bibliothek mit etwa 2.000 Verbindungen - für eine Reihe verschiedener Konfigurationen der Mpro-Protease des SARS-CoV-2-Virus durchgeführt, die durch eine Molekulardynamikstudie charakterisiert wurden.
Daher wird in dieser Arbeit zunächst das dynamische Profil der Mpro-Protease in der apo-Form durch konventionelle (cMD) und Gauß-beschleunigte Molekulardynamiksimulationen (GaMD) charakterisiert und eine Reihe repräsentativer Strukturen definiert. Diese Strukturen wurden dann zur Durchführung der molekularen Assemblierung verwendet, einer bioinformatischen Methode, mit der die günstigste Position der Wechselwirkung zwischen einem Liganden und seinem Zielprotein vorhergesagt und mit Hilfe von Computertechniken berechnet werden kann. Anschließend wurde ein iterativer Prozess entwickelt, um die Länge der cMDs der Protein-Ligand-Komplexe zu erhöhen und die freie Bindungsenergie zu berechnen, um die vielversprechendsten Kandidaten auszuwählen.
Auf der Grundlage der Ergebnisse wurden elf Verbindungen ausgewählt und in vitro auf ihre Fähigkeit zur Hemmung der Mpro-Protease getestet. Schließlich wurden aus der Naturstoffdatenbank fünf Kandidaten für antivirale Mpro-Protease-Inhibitoren für SARS-CoV-2 identifiziert.
Hinweis: Dieser Artikel wurde mit einem Computersystem ohne menschlichen Eingriff übersetzt. LUMITOS bietet diese automatischen Übersetzungen an, um eine größere Bandbreite an aktuellen Nachrichten zu präsentieren. Da dieser Artikel mit automatischer Übersetzung übersetzt wurde, ist es möglich, dass er Fehler im Vokabular, in der Syntax oder in der Grammatik enthält. Den ursprünglichen Artikel in Spanisch finden Sie hier.