Naturstoffschätze im Organismen-Dreieck
Umfassende Analyse der Genomsequenzen insektenpathogener Bakterien offenbart eine Fülle einzigartiger bioaktiver Naturstoffe
Die Wechselwirkungen zwischen Mikroben und anderen Organismen werden durch eine Vielzahl kleiner Moleküle vermittelt, die man auch als Naturstoffe bezeichnet. Ein Forscherteam unter der Leitung von Dr. Yi-Ming Shi und Prof. Helge Bode vom MPI für terrestrische Mikrobiologie hat nun eine systematische Analyse dieser unbekannten biochemischen „dunklen Materie" und ihrer biologischen Funktionen durchgeführt. Die Arbeit schafft eine solide Grundlage für die künftige Entdeckung und Verbesserung von Naturstoffen.
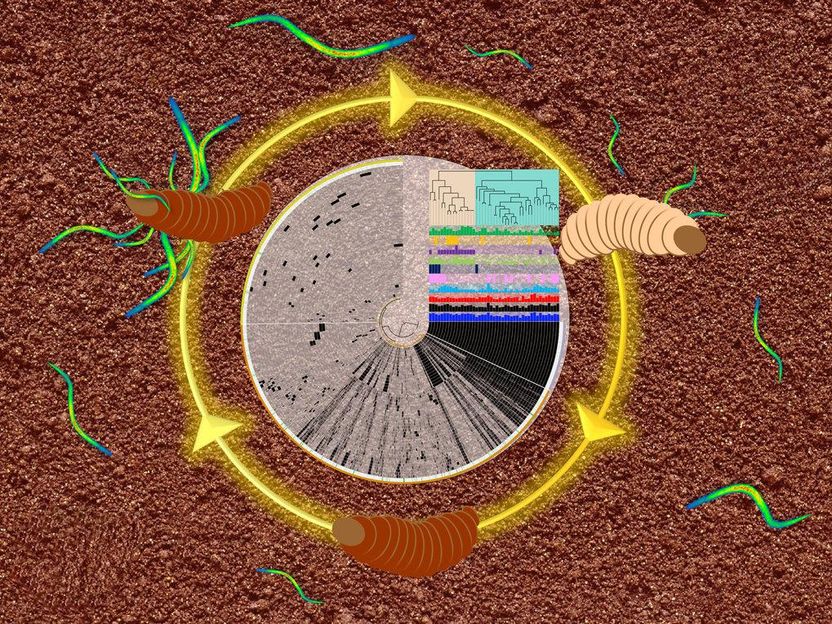
Lebenszyklus der Nematoden-Bakterien- Gemeinschaft und Darstellung des Pangenoms der analysierten Xenorhabdus und Photorhabdus-Bakterien. Bakterien der Gattungen Photorhabdus und Xenorhabdus leben als Symbionten im Darm von bodenbewohnenden Nematoden der Gattungen Heterorhabditis und Steinernema. Die Nematoden infizieren Insektenlarven, in die sie die Bakterien injizieren. Die Bakterien wiederum töten die Insektenlarven, die dann eine Nahrungsquelle für Bakterien und Nematoden darstellen. Trotz dieser Komplexität lässt sich dieses dreigliedrige Modellsystem im Labor in verschiedenen Teilkombinationen und in allen Lebensstadien untersuchen.
© Max Planck Institute for Terrestrial Microbiology/Shi
In Wechselwirkungen von Bakterien mit anderen Organismen kommt eine Myriade von Naturstoffen zum Einsatz. In den meisten Fällen lässt sich nicht nachverfolgen, welcher Bakterienstamm welchen Naturstoffe bildet und welche Eigenschaften dieser hat. Daher sind bekannte Stoffe wie der Giftstoff Colibactin, der aus menschlichen Darmbakterien stammt, eher die Ausnahme.
Insektenpathogene Bakterien: Forschungsmodelle mit großem Potential
Xenorhabdus und Photorhabdus leben in Symbiose mit spezifischen Fadenwürmern (Nematoden). Mit ihnen gemeinsam infizieren und töten sie bodenlebende Insektenlarven. Innerhalb dieser Beziehung sind die Bakteriengattungen für die Produktion zahlreicher Giftstoffe (Toxine) und anderer interaktionsvermittelnder Stoffe verantwortlich. Gleichzeitig lassen sie sich im Labor auch ohne ihren Nematoden-Wirt züchten und genetisch manipulieren. Daher gelten sie als vielversprechende Forschungsmodelle für die Untersuchung der chemischen Ökologie bakterieller Naturstoffe.
Nun wurden die Bakterien in einer Kombination aus pangenomischer und Domänen-genomischer Analyse erstmals systematisch und im Hinblick auf zwei zentrale Fragen untersucht: welche Gene oder Gencluster sind für die Naturstoffproduktion verantwortlich, wie weit verbreitet oder einzigartig sind diese Gene in den verschiedenen Bakteriengattungen?
Zahlreiche unbekannte Biosynthese-Gencluster entdeckt
Das Forscherteam unter der Leitung von Dr. Yi-Ming Shi und Prof. Dr. Helge Bode vom Max-Planck-Institut für terrestrische Mikrobiologie identifizierte 1.000 Biosynthese-Gencluster, von denen über die Hälfte bisher unbekannt waren. Die Anwendung von homologer Expression, chemischer Strukturaufklärung und biologischer Assays offenbarte dem Forscherteam eine wahre biochemische Schatzkammer. Aber wie profitierten die Nematoden als Bakterienwirte von diesen bakteriellen Stoffen?
„Unsere Analysen der Bioaktivität zeigt, dass es sich bei den am weitesten verbreiteten Naturstoffen um eukaryotische Proteasom-Inhibitoren handelt. Diese unterdrücken vermutlich das Immunsystem von Insekten. Aber wir fanden auch andere Virulenzfaktoren gegen Insekten, oder Immunsuppressoren. So liefern unsere Ergebnisse wichtige Erkenntnisse zur Funktion dieser bakteriellen Naturstoffe", sagt Yi-Ming Shi, Erstautor der Publikation, die im Fachmagazin „Nature Chemistry“ erschienen ist. "Viele unserer Neuentdeckungen stechen nicht nur durch ihre ungewöhnlichen chemischen Strukturmerkmale und Wirkungsweisen hervor, sondern auch durch ihr Vorkommen in verschiedenen Bakterientaxa. Dies deutet auf mögliche universelle chemische Waffen in Bakterien hin.“
Bakterien mit besonderer Fähigkeit zur Naturstoffproduktion
Die Identifizierung und funktionelle Charakterisierung der Naturstoffe fanden in Kooperation mit Prof. Yonggyun Kim von der Andong National University (Korea), Prof. Michael Groll von der Technischen Universität München und Prof. Rolf Müller vom Helmholtz Institut für Pharmazeutische Forschung Saarland statt. Die Ergebnisse zeigten, wie die Bakterien das Zusammenspiel der Naturstoffe zur Aufrechterhaltung der symbiotischen Lebensweise orchestrieren, und wiesen gleichzeitig auf die enormen Fähigkeiten insbesondere der Gattung Xenorhabdus hin, neuartige Naturstoffe zu produzieren. Damit schafft diese Arbeit auch wichtige Voraussetzungen, um die Entdeckung und mögliche Anwendung von Naturstoffen in Mikrobiomen aus anderen Nischen deutlich zu beschleunigen.
Originalveröffentlichung
Shi, Y.-M.; Hirschmann, M.; Shi, Y-N.; Ahmed, S.; Abebew, D.; Tobias, N.J.; Grün, P.; Crames, J.P.; Pöschel, L.; Kuttenlochner, W.; Richter, C.; Herrmann, J.; Müller, R.; Thanwisai, A; Pidot, S. J.; Stinear, T. P.; Groll, M.; Kim, Y.; Bode, H. B.; Global analysis of biosynthetic gene clusters reveals conserved and unique natural products in entomopathogenic nematode-symbiotic bacteria; Nature Chemistry 14 (2022)

























































