Nachweis von Krebs-Biomarkern aus Blutproben mittels DNA-Computing-Technologie auf Nanoporenbasis
Neue Methode zum Nachweis von miRNA-Krebsmustern entwickelt
Das Cholangiokarzinom, auch bekannt als Gallengangskrebs, ist eine Krebsart mit einer charakteristisch hohen Sterblichkeit. Zum Zeitpunkt der Diagnose sind die meisten Gallengangskarzinome in der Regel bereits unheilbar. Deshalb sind Methoden zur Früherkennung von Gallengangskrebs dringend erforderlich.
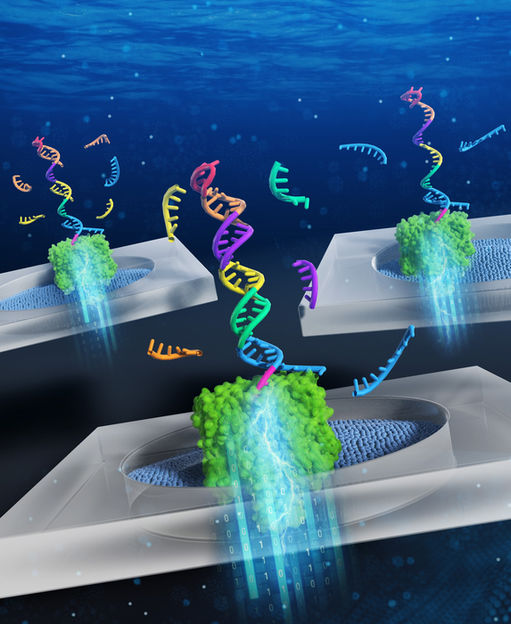
Mustererkennung der miRNA-Expression mit Hilfe von DNA-Computing und Nanopore-Dekodierung
Ryuji Kawano, Tokyo University of Agriculture and Technology
Die Flüssigbiopsie, die Entnahme von nicht festem biologischem Gewebe wie Blut, gewinnt als schnelle und nicht-invasive Methode zur Diagnose von Krebserkrankungen zunehmend an Interesse. Im Gegensatz zu herkömmlichen Biopsien, die einen chirurgischen Eingriff und oft auch eine Vollnarkose erfordern, werden für eine Flüssigblutbiopsie nur einige Milliliter Blut benötigt, was für den Patienten mit minimalen Risiken verbunden ist.
Nach der Entnahme wird das Blut auf spezifische Marker untersucht, die auf das Vorhandensein von Krebsgewebe hinweisen. So werden beispielsweise bestimmte Muster von microRNA (miRNA), kurzen, nicht codierenden RNA-Strängen, mit verschiedenen Krebsarten in Verbindung gebracht und können dazu verwendet werden, Krebserkrankungen anhand von Flüssigbiopsien mit hoher Präzision zu diagnostizieren. Aufgrund der geringen Konzentration von miRNA in Blutproben ist ihr Nachweis jedoch schwierig.
Forscher der Universität für Landwirtschaft und Technologie in Tokio haben eine neue Methode zum Nachweis von miRNA-Krebsmustern entwickelt, die auf der DNA-Computertechnologie basiert. Die entwickelte Methode erweist sich als vielversprechendes Instrument für eine einfache und frühzeitige Krebsdiagnose aus Flüssigbiopsien mit niedrigen Konzentrationen der Zielbiomarker. Die Ergebnisse wurden am 26. Juni 2022 in der von Experten begutachteten Fachzeitschrift JACS Au veröffentlicht.
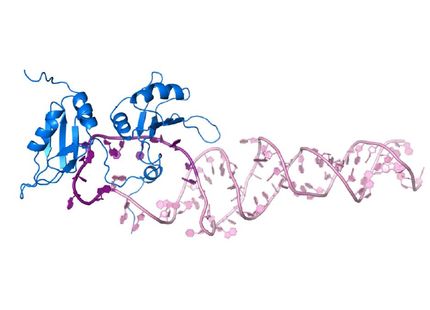
"DNA-Computing nutzt die biochemischen Reaktionen der informationskodierenden DNA-Moleküle, um Probleme auf der Grundlage formaler Logik zu lösen, so wie es auch normale Computer tun", sagte der korrespondierende Autor Ryuji Kawano, Professor an der Tokyo University of Agriculture and Technology (TUAT) in Japan. "In diesem Fall wurde ein diagnostisches DNA-Molekül entwickelt, das in der Lage ist, fünf verschiedene Arten von miRNA zu binden, die mit Gallengangskrebs in Verbindung stehen. Bei der Bindung der miRNA-Moleküle wandelt die diagnostische DNA das Expressionsmuster der miRNAs in die Information um, die in Form einer Nukleinsäurestruktur enthalten ist."
Um diese Information auszulesen, verwenden die Wissenschaftler eine Methode namens Nanopore-Decodierung. Bei dieser Methode wird die DNA durch ein Loch in Nanogröße, eine "Pore", geleitet. Wenn das Molekül die Pore durchquert, behindert es den Fluss des elektrischen Stroms durch die Pore. Diese Störungen des Stroms durch die Pore können dann gemessen und dazu verwendet werden, die Eigenschaften des durchgelassenen Moleküls abzuleiten. Im Falle der diagnostischen DNA werden die gebundenen miRNAs von der DNA "abgelöst", was zu einer Stromhemmung mit charakteristischer Amplitude und Dauer führt. Durch die statistische Analyse der Unzipping-Daten der miRNA-Muster waren die Wissenschaftler in der Lage, krebsspezifische Expressionsmuster selbst aus klinischen Proben mit extrem niedrigen miRNA-Konzentrationen zu erkennen. Dies ist ein bedeutender Fortschritt auf dem Gebiet der Nanoporen-Diagnose, da Nanoporen-Messungen im Allgemeinen als nicht in der Lage angesehen wurden, Nukleinsäuren in so geringen Konzentrationen nachzuweisen, was den Einsatz der Technologie in klinischen Anwendungen untergraben hat.
Hinweis: Dieser Artikel wurde mit einem Computersystem ohne menschlichen Eingriff übersetzt. LUMITOS bietet diese automatischen Übersetzungen an, um eine größere Bandbreite an aktuellen Nachrichten zu präsentieren. Da dieser Artikel mit automatischer Übersetzung übersetzt wurde, ist es möglich, dass er Fehler im Vokabular, in der Syntax oder in der Grammatik enthält. Den ursprünglichen Artikel in Englisch finden Sie hier.