Neues Protokoll erlaubt Analyse von Stoffwechselprodukten aus fixiertem Gewebe
Wissenschaftler am Helmholtz Zentrum München haben eine neue Methode für die bildgebende Massenspektrometrie entwickelt, mit der es erstmals möglich ist, in fixierten Gewebeproben hunderte von Metaboliten gleichzeitig zu analysieren. Ihre Publikation in ‚Nature Protocols‘ erklärt den neuen Zugang zu den Stoffwechselinformationen, der ein bislang nicht ausgeschöpftes Potenzial für gewebsbasierte Forschung und Molekulare Diagnostik bietet.
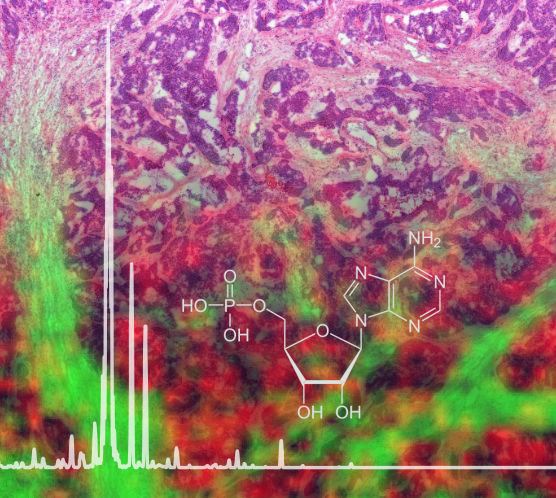
Ein neues Protokoll für die bildgebende Massenspektrometrie erlaubt die Analyse von Stoffwechselprodukten wie Adenosinmonophosphat aus fixiertem Gewebe, hier im Hintergrund dargestellt.
Helmholtz Zentrum München
In der biomedizinischen Forschung ist das Arbeiten mit Gewebeproben nicht mehr wegzudenken, denn sie erlauben abseits von Petrischale und Computersimulation einen Blick in die biologische Wirklichkeit, etwa von Patienten. Um das Gewebe für spätere Untersuchungen möglichst im Originalzustand aufzubewahren, wird es in der Regel in Formalin fixiert und in wachsartiges Paraffin eingebettet.
Bisher war man davon ausgegangen, dass in so behandeltem Material eine Analyse von Stoffwechselprodukten (Metaboliten) im Gegensatz zu DNA oder Proteinen aus technischen Gründen kaum möglich ist. Dies konnte ein Wissenschaftlerteam der Abteilung Analytische Pathologie des Helmholtz Zentrums München um Leiter Prof. Dr. Axel Karl Walch nun widerlegen.
Fixiertes Gewebe im großen Maßstab zugänglich
Die Forscherinnen und Forscher entwickelten ein Protokoll, wonach es binnen eines Tages möglich ist, die Metabolitkomposition eines Gewebes mit Hilfe der bildgebenden Massenspektrometrie zu bestimmen und in Gewebeschnitten sichtbar zu machen. Dazu reichen den Autoren zufolge relativ kleine Mengen an Material. „Unsere Methode erlaubt auch die Analyse von kleinsten Biopsien und sogar Gewebe-Microarrays, was sie für die molekulare Forschung und Diagnostik besonders interessant macht“, erklärt Doktorand Achim Buck, gemeinsam mit Alice Ly, Erstautor der Studie.
Um auszuschließen, dass die gemessenen Daten nicht durch den Fixationsprozess verfälscht werden, verglichen die Autoren sie mit Messwerten der gleichen Proben, die aber nicht fixiert, sondern schockgefroren waren. „Ein Großteil der gemessenen Metabolite fand sich in beiden Analysen wieder“, berichtet Achim Buck. „Wir konnten zeigen, dass die Methode verlässlich funktioniert und dabei die aufwändige Logistik und Lagerung von schockgefrorenen Proben umgeht.“
Neben der einfachen Handhabung und der hohen Reproduzierbarkeit ist den Wissenschaftlern zufolge auch die Möglichkeit, mit hohem Probendurchsatz zu arbeiten, ein wichtiger Vorteil der neuen Methode.* Vor allem aber könne man nun die räumliche Verteilung von Molekülen im Gewebe bildhaft und mit großer Präzision studieren. „Das ist sowohl in der Forschung als auch in der klinisch diagnostischen Praxis ein enormer Vorteil“, ordnet Studienleiter Walch die neuen Möglichkeiten ein. „Unser Ziel ist es nun, mit unserem neuen Analyseverfahren zukünftig neue prädiktive, diagnostische und prognostische Marker in Geweben zu identifizieren, sowie Krankheitsprozesse besser zu verstehen.“
Von der Veröffentlichung des Protokolls erhoffen sich die Wissenschaftler auch einen Austausch und eine Weiterentwicklung durch Kollegen, um metabolische Untersuchungen an Archivgeweben voranzutreiben.
Originalveröffentlichung
Alice Ly, Achim Buck, Benjamin Balluff, Na Sun, Karin Gorzolka, Annette Feuchtinger, Klaus-Peter Janssen, Peter J K Kuppen, Cornelis J H van de Velde, Gregor Weirich, Franziska Erlmeier, Rupert Langer, Michaela Aubele, Horst Zitzelsberger, Liam McDonnell, Michaela Aichler & Axel Walch; "High-mass-resolution MALDI mass spectrometry imaging of metabolites from formalin-fixed paraffin-embedded tissue"; Nature Protocols; 2016
Verwandte Inhalte finden Sie in den Themenwelten
Themenwelt Massenspektrometrie
Die Massenspektrometrie ermöglicht es uns, Moleküle aufzuspüren, zu identifizieren und ihre Struktur zu enthüllen. Ob in der Chemie, Biochemie oder Forensik – Massenspektrometrie eröffnet uns ungeahnte Einblicke in die Zusammensetzung unserer Welt. Tauchen Sie ein in die faszinierende Welt der Massenspektrometrie!
Themenwelt Massenspektrometrie
Die Massenspektrometrie ermöglicht es uns, Moleküle aufzuspüren, zu identifizieren und ihre Struktur zu enthüllen. Ob in der Chemie, Biochemie oder Forensik – Massenspektrometrie eröffnet uns ungeahnte Einblicke in die Zusammensetzung unserer Welt. Tauchen Sie ein in die faszinierende Welt der Massenspektrometrie!





















































