Fortschritt in der DNA-Analyse
Die Arbeitsgruppe um Prof. Daniel Summerer an der Fakultät für Chemie und Chemische Biologie der TU Dortmund hat ein neuartiges Verfahren entwickelt, das die Suche nach epigenetisch modifizierten Genabschnitten für die Forschung vereinfacht. Das Besondere an dieser Methode ist, dass sich dank eines Bauplans für maßgeschneiderte Proteinsonden gezielt jede beliebige DNA-Sequenz auf etwaige Modifikationen untersuchen lässt - und das mit hoher Genauigkeit. Solche Nachweisverfahren sind insbesondere für die Krebsdiagnostik relevant, da sie zeigen, welche Gene in den Tumorzellen eines Patienten infolge der Modifikation ein- oder ausgeschaltet sind und welche Therapien wirken könnten.
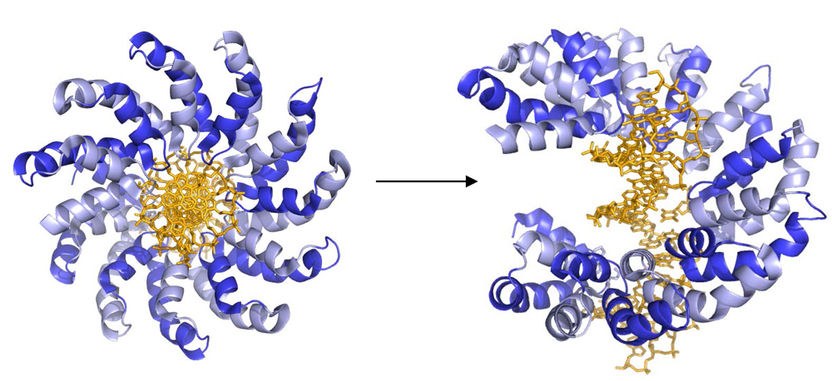
TALES (blau) binden an bestimmte Sequenzen der DNA (gelb), damit diese genauer analysiert werden können.
Prof. Daniel Summerer/TU Dortmund
Es ist schon lange bekannt, dass Mutationen im Genom Krebs auslösen können. Doch nicht nur ein falscher DNA-Baustein kann die Ursache für die Entstehung eines Tumors sein. Auch außerplanmäßig eingefügte oder fehlende epigenetische Modifikationen der korrekten Bausteine können das Zellwachstum stören und eine gesunde Zelle in eine wuchernde Krebszelle verwandeln. Aus diesem Grund interessieren sich Prof. Summerer und sein Team für solche Modifikationen von DNA-Sequenzen, insbesondere für die Methylierung und abgeleitete Modifikationen des DNA-Bausteins Cytosin, durch die eine Zelle ihre Gene flexibel ein- oder ausschalten kann.
Die herkömmliche DNA-Analyse liest normalerweise jedoch nur die Sequenz der vier DNA-Bausteine (neben Cytosin noch Adenin, Guanin und Thymin) aus, nicht auch deren Modifikationen – dafür muss man Tricks anwenden. Die Arbeitsgruppe von Prof. Summerer hat hier von der Natur abgeguckt: In bestimmten Erregern von Pflanzenkrankheiten gibt es Proteine, die flexibel an Gene in Zellen der Wirtspflanze binden und diese regulieren können, was im sogenannten Genom-Engineering bereits genutzt wird.
Die Arbeitsgruppe hat die Selektivitätsmechanismen dieser Proteine „gehackt“ und einen Bauplan für Proteinsonden mit einer erweiterten Programmierbarkeit der DNA-Erkennung entwickelt. So kann sie inzwischen TALE-Proteine (transcription-activator like effector proteins) derart programmieren, dass diese spezifisch an die zu untersuchende DNA-Sequenz binden und dabei zwischen unterschiedlichen modifizierten und der nicht-modifizierten Cytosin-Variante unterscheiden können.
Mithilfe der TALE-Proteine lassen sich nun erstmals beliebige modifizierte und nicht-modifizierte DNA-Abschnitte aus Tumorproben im Labor gezielt und direkt voneinander trennen, bevor es zur weiteren Analyse geht. Das vorliegende Verhältnis von DNA mit modifiziertem und nicht-modifiziertem Baustein ist charakteristisch für die Art des Tumors und kann zum Beispiel Hinweise darauf geben, welche Medikamente helfen und welche nicht. So ist beispielsweise bekannt, dass bei der häufigsten Leukämieform von Erwachsenen eine Chemotherapie oft nicht erforderlich ist, sofern in einem bestimmten Gen ein Cytosin methyliert ist, während der umgekehrte Fall eine sofortige Therapie notwendig macht.
Der Nachweis von derlei DNA-Modifikationen war bisher schon durch andere Tricks im Labor möglich, jedoch erlauben bisherige Verfahren keine direkte Trennung von frei wählbaren, modifizierten und nicht-modifzierten DNA Abschnitten wie die neue Methodik aus Dortmund. Die Ergebnisse wurden kürzlich in der Fachzeitschrift Journal of the American Chemical Society (JACS) veröffentlicht.
Meistgelesene News
Weitere News aus dem Ressort Wissenschaft

Holen Sie sich die Analytik- und Labortechnik-Branche in Ihren Posteingang
Mit dem Absenden des Formulars willigen Sie ein, dass Ihnen die LUMITOS AG den oder die oben ausgewählten Newsletter per E-Mail zusendet. Ihre Daten werden nicht an Dritte weitergegeben. Die Speicherung und Verarbeitung Ihrer Daten durch die LUMITOS AG erfolgt auf Basis unserer Datenschutzerklärung. LUMITOS darf Sie zum Zwecke der Werbung oder der Markt- und Meinungsforschung per E-Mail kontaktieren. Ihre Einwilligung können Sie jederzeit ohne Angabe von Gründen gegenüber der LUMITOS AG, Ernst-Augustin-Str. 2, 12489 Berlin oder per E-Mail unter widerruf@lumitos.com mit Wirkung für die Zukunft widerrufen. Zudem ist in jeder E-Mail ein Link zur Abbestellung des entsprechenden Newsletters enthalten.