Moleküle bewegen mit Fingerspitzengefühl
Neue Methode in der Rastertunnelmikroskopie: Forscher bilden Schriftzug aus 47 Molekülen
Jülicher Wissenschaftler haben eine neue Ansteuerung für Rastertunnelmikroskope entwickelt, die es ihnen ermöglicht, einzelne große Moleküle interaktiv mit der Hand zu verschieben. Bisher waren nur simple, starr programmierte Bewegungen möglich. Zum Test haben die Forscher aus einer einlagigen Molekülschicht einen Schriftzug herausgetrennt, der aus gerade einmal 47 Molekülen besteht. Das Verfahren eröffnet grundlegend neue Möglichkeiten für die Konstruktion von molekularen Transistoren und anderen Nano-Bauteilen. Die Ergebnisse sind in der Fachzeitschrift Beilstein Journal of Nanotechnology erschienen.

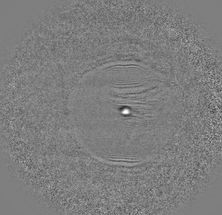
Schriftzug aus nur 47 Molekülen
Forschungszentrum Jülich
"Mit dem Verfahren lassen sich erstmalig auch große organische Moleküle kontrolliert aus zusammenhängenden Strukturen herausgreifen und neu platzieren", erläutert Dr. Ruslan Temirov vom Jülicher Peter Grünberg Institut. Damit sind die Wissenschaftler einen Schritt weiter gekommen bei der Suche nach einer Technik, mit der sich einzelne Moleküle frei zu komplexen Strukturen zusammenfügen lassen. Ein solches Bausteinsystem für die Nanotechnologie wird von Forschergruppen weltweit verfolgt und gilt als entscheidende Voraussetzung für die Entwicklung neuartiger elektronischer Bauteile der nächsten Generation.
Mittels Bewegungserkennung haben die Wissenschaftler der von Temirov geleiteten Nachwuchsgruppe die Hand des Operators direkt mit dem Rastertunnelmikroskop gekoppelt. Mit dessen Spitze lassen sich die Moleküle anheben und wieder absetzen wie mit einem Kran. Mit einer Übersetzung von Fünfhundert Millionen zu Eins werden die groben menschlichen Bewegungen dabei in atomare Dimensionen übertragen. "Eine Verschiebung der Hand um fünf Zentimeter bewirkt, dass sich die feine Spitze des Rastertunnelmikroskops gerade einmal um einen Angström über die Probe bewegt. Das entspricht der typischen Größenordnung von Atomradien und Bindungslängen in Molekülen", erklärt Ruslan Temirov.
Die Steuerung braucht allerdings etwas Übung. "Der erste Versuch, ein Molekül abzulösen, dauerte noch 40 Minuten. Danach waren es nur noch um die 10 Minuten", berichtet Matthew Green. Insgesamt vier Tage brauchte der Doktorand, um 47 Moleküle zu entfernen und damit das Wort "JÜLICH" in eine einlagige Schicht Perylentetracarbonsäuredianhydrid (PTCDA) zu schreiben. Dabei handelt es sich um einen organischen Halbleiter, der eine wichtige Rolle spielt bei der Entwicklung organischer Elektronik, mit der sich etwa biegsame Bauelemente oder günstige Einweg-Chips zum Aufdrucken realisieren lassen, die mit konventioneller Silizium-Technologie undenkbar sind.
Sogar kleine "Schreibfehler" ließen sich mit der neuen Methode problemlos korrigieren. Ein versehentlich herausgegriffenes Molekül beim Querstrich des "H" konnte Green einfach durch ein neues Molekül ersetzen, das er vom Rand der Schicht abgelöst hatte. "Genau hier zeigt sich der Vorteil der Methode. Der Experimentator kann in den Vorgang eingreifen und nach einer Lösung suchen, wenn sich ein Molekül versehentlich löst oder ungeplant wieder an seine ursprüngliche Position zurückspringt", so der Physiker.
Die interaktive Vorgehensweise ermöglicht es insbesondere, Moleküle kontrolliert zu verschieben, die sich in größeren zusammenhängenden Strukturen befinden. Im Gegensatz zu einzelnen Atomen und Molekülen, deren Bewegung mit dem Rastertunnelmikroskop längst wissenschaftliche Routine ist, ließen sich solche größere Molekülverbünde bisher kaum gezielt bearbeiten. Denn die Bindungskräfte von Molekülen, die ringsum an ihre Nachbarmoleküle gebunden sind, lassen sich kaum exakt vorhersagen. Erst während des Experiments zeigt sich daher, welche Kraft zum Ablösen nötig ist und über welchen Pfad sich das Molekül ohne Komplikationen entfernen lässt.
Die gesammelten Erfahrungen sollen dazu beitragen, die zeitraubenden Arbeitsschritte zu erleichtern. "In Zukunft soll ein selbstlernender Computer die aufwendige Molekül-Manipulation übernehmen. Die für dieses Projekt notwendige Intuition für Nano-Mechanik gewinnen wir jetzt durch unsere neuartige Steuerung und sprichwörtlich in Handarbeit", so Dr. Christian Wagner, der ebenfalls der Jülicher Arbeitsgruppe angehört.