Auf dem Weg zu Biosensoren mit Graphen
Erstmals ist es einem Team gelungen, nicht nur präzise zu messen, sondern sogar zu steuern, wie stark eine Graphenschicht eine organische Verbindung absorbiert. Dies könnte in Zukunft ermöglichen, Graphen als empfindlichen Sensor für Biomoleküle zu nutzen.
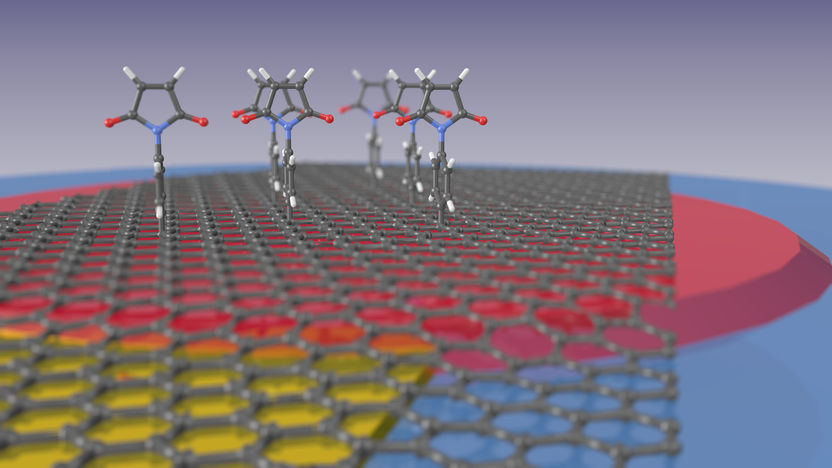
Die Zeichnung veranschaulicht wie Maleimid-Verbindungen an der Graphenoberfläche andocken. Dabei liegt die Graphen-Monolage auf einer dünnen Schicht aus Siliziumnitrid (rot) auf einer Quarzmikrowaage (blau) und kann mit einem Goldkontakt (gelb) unter Spannung gesetzt werden.
Marc A. Gluba/HZB
Reiner Kohlenstoff tritt in vielfältiger Gestalt auf: zu den klassischen Strukturen von Diamant, Graphit und Kohle sind in letzter Zeit auch exotischere Geschwister dazugekommen, zum Beispiel Graphen. Die Struktur ähnelt einer Bienenwabe, sie besteht aus sechseckigen Maschen, an deren Ecken stets ein Kohlenstoffatom sitzt und ist nur eine einzige Atomschicht dick, also quasi zweidimensional. Dabei ist Graphen extrem leitfähig, völlig transparent und mechanisch wie chemisch äußerst belastbar.
Graphen ist bislang nicht sehr wählerisch
Dass Graphen sich grundsätzlich auch als hochempfindlicher Sensor zum Nachweis organischer Moleküle eignet, ist schon länger bekannt. Denn sobald fremde Moleküle andocken, sinkt die elektrische Leitfähigkeit des Graphens. Das Problem ist nur: Das passiert bei fast jedem Molekül, Graphen ist also nicht sehr selektiv, so dass unterschiedliche Moleküle nicht zu unterscheiden sind. So ist es als Sensor nicht zu gebrauchen.
Jetzt: Halterungen für "Schloss-Moleküle" angebracht
Nun hat ein Team vom HZB-Institut für Silizium-Photovoltaik einen interessanten Weg beschritten, um die Selektivität zu erhöhen: Es gelang ihnen, Graphen elektrochemisch zu funktionalisieren und für die Aufnahme von Sonden-Molekülen vorzubereiten. Dafür wurden aus einer organischen Lösung über dem Graphen para-Maleimidophenyl-Gruppen auf die Graphen-Oberfläche aufgebracht. Diese organischen Moleküle funktionieren wie Halterungen, an die im nächsten Schritt die Sonden-Moleküle angebracht werden können. „Aufgrund dieser Moleküle kann das Graphen nun, ähnlich dem Schlüssel-Schloss-Prinzip, zur Detektion von verschiedensten Stoffen verwendet werden“ erklärt Dr. Marc Gluba. Die „Schloss“-Moleküle auf der Oberfläche sind hoch selektiv und nehmen ausschließlich die passenden „Schlüssel“-Moleküle auf.
Großflächige Graphenproben am HZB
Auch andere Forschungsgruppen hatten schon in dieser Richtung Versuche angestellt, allerdings standen ihnen nur winzig kleine Graphenflöckchen mit Durchmessern im Mikrometerbereich zur Verfügung, so dass von den Rändern ausgelöste Effekte dominierten. Am HZB haben Physiker und Chemiker dagegen Graphenflächen von mehreren Quadratzentimetern hergestellt, so dass Randeffekte im Vergleich zu den Prozessen in der Fläche kaum noch eine Rolle spielen. Die Graphenschicht brachten sie auf einer Quarzmikrowaage auf. Jede Massenzunahme verändert dabei die Schwingfrequenz des Quarzkristalls, wodurch kleinste Massen bis hin zu Einzelmoleküllagen messbar werden.
Präzise Messung und Steuerung
„Wir konnten damit erstmals präzise und quantitativ nachweisen, wie viele Moleküle tatsächlich auf der Oberfläche des Graphens aufgebracht wurden“, berichtet der Nachwuchsforscher Felix Rösicke, der diese Frage für seine Doktorarbeit untersucht hat. „Mit Hilfe einer angelegten Spannung können wir darüber hinaus genau steuern, wie viele Moleküle am Graphen andocken“, erklärt Dr. Jörg Rappich vom HZB-Institut für Silizium-Photovoltaik, der Rösicke betreut.
„Die Hoffnungen, die sich mit Graphen verbinden, sind wirklich fantastisch“, sagt Prof. Dr. Norbert Nickel, Leiter der Arbeitsgruppe. Als Vision könne man sich zum Beispiel ein wirklich preisgünstiges „Lab on a Chip“ vorstellen, in das man einen einzigen Blutstropfen gibt, und sofort Werte für medizinisch interessante Parameter etc. erhält.
Originalveröffentlichung
Originalveröffentlichung
Quantifying the electrochemical maleimidation of large area graphene; F. Rösicke, M.A. Gluba, K. Hinrichs, Guoguang Sun, N.H. Nickel, J. Rappich; Electrochemistry Communications; Volume 57, August 2015, Pages 52–55
Organisationen
Weitere News aus dem Ressort Wissenschaft
Diese Produkte könnten Sie interessieren

Octet RH16 and RH96 von Sartorius
Effiziente Proteinanalyse im Hochdurchsatz zur Prozessoptimierung und Herstellungskontrolle
Markierungsfreie Protein-Quantifizierung und Charakterisierung von Protein-Protein Wechselwirkungen

Octet SF3 von Sartorius
Molekulare Bindungskinetik und Affinität mit einer einzigen dynamischen SPR-Injektion
Die Kurvenkrümmung ist der Schlüssel akkurater biomolekularer Wechselwirkungsanalyse

Octet R2 / Octet R4 / Octet R8 von Sartorius
Vollgas auf 2, 4 oder 8 Kanälen: Molekulare Wechselwirkungen markierungsfrei in Echtzeit analysieren
Innovative markierungsfreie Echtzeit-Quantifizierung, Bindungskinetik und schnelle Screening-Assays

Holen Sie sich die Analytik- und Labortechnik-Branche in Ihren Posteingang
Mit dem Absenden des Formulars willigen Sie ein, dass Ihnen die LUMITOS AG den oder die oben ausgewählten Newsletter per E-Mail zusendet. Ihre Daten werden nicht an Dritte weitergegeben. Die Speicherung und Verarbeitung Ihrer Daten durch die LUMITOS AG erfolgt auf Basis unserer Datenschutzerklärung. LUMITOS darf Sie zum Zwecke der Werbung oder der Markt- und Meinungsforschung per E-Mail kontaktieren. Ihre Einwilligung können Sie jederzeit ohne Angabe von Gründen gegenüber der LUMITOS AG, Ernst-Augustin-Str. 2, 12489 Berlin oder per E-Mail unter widerruf@lumitos.com mit Wirkung für die Zukunft widerrufen. Zudem ist in jeder E-Mail ein Link zur Abbestellung des entsprechenden Newsletters enthalten.