Mit Bakterien infizierte Zellen einzeln analysieren
Die Reaktion einzelner Zellen auf eine Infektion mit Bakterien analysieren: Das geht mit einer neuen Technik, die ein Würzburger Forschungsteam präsentiert. Sie gewährt auch gleich überraschende neue Einblicke in die Welt der Salmonellen.
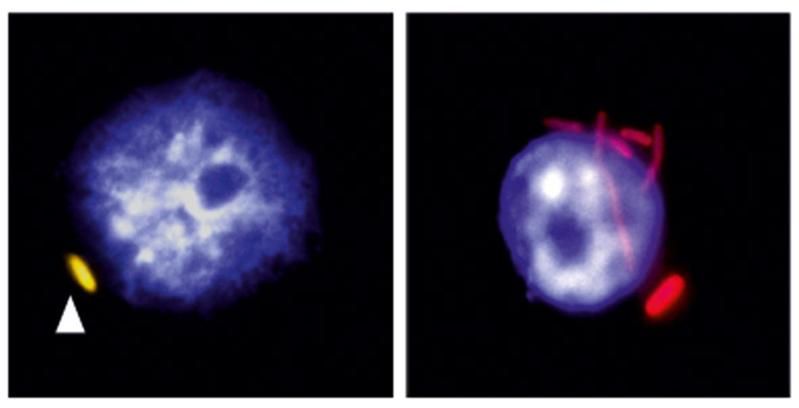
Links ein Makrophage (Zellkern in blau), in dem eine Salmonelle (gelb) ruht. Rechts ein Makrophage, in dem sich die Bakterien (rot) vermehrt haben.
Antoine-Emmanuel Saliba
Infektionskrankheiten zählen nach wie vor zu den weltweit häufigsten Todesursachen. Um neue Therapiemöglichkeiten oder Impfungen zu entwickeln, will die Wissenschaft noch besser als bisher verstehen, wie Viren, Pilze und Bakterien den Menschen krank machen.
Ein Spezialfall dabei sind Salmonellen und andere Bakterien, die in die Zellen des Menschen eindringen können und sich dort vermehren. Hier richtet die Forschung ihren Blick in jüngster Zeit verstärkt auch auf die Wirtszellen und die individuellen Unterschiede, die es zwischen ihnen gibt.
Für einen Fortschritt auf diesem Gebiet sorgt nun eine Forschungsgruppe der Universität Würzburg: Sie hat eine neue Technik entwickelt, mit der sich die Wechselwirkungen einzelner Wirtszellen mit Bakterien analysieren lassen. Die Ergebnisse sind im Fachjournal „Nature Microbiology“ veröffentlicht. Sie stammen von Professor Jörg Vogel und seiner Gruppe vom Institut für Molekulare Infektionsbiologie, aus der Core Unit Systemmedizin der Medizinischen Fakultät und aus dem Imperial College in London.
Salmonellen verfolgen zwei Strategien
Das Team hat mit der Technik namens „Single-cell RNA-seq“ die Infektion von Makrophagen mit Salmonellen studiert. Makrophagen sind Zellen des Immunsystems, die zu den weißen Blutkörperchen gehören. Bei Salmonellen handelt es sich um krankheitserregende Bakterien, die über kontaminiertes Essen oder Wasser aufgenommen werden können. Sie verursachen in der Regel Magen-Darm-Entzündungen und Durchfall. Bei Menschen mit geschwächtem Immunsystem können sie aber den ganzen Organismus überfluten und dann lebensbedrohlich werden.
Wenn sie erst einmal in eine Makrophage eingedrungen sind, verfolgen Salmonellen zwei Strategien. Entweder vermehren sie sich dort sehr stark – oder sie verfallen in einen Zustand, der es ihnen erlaubt, über Jahre hinweg in der Zelle und im Wirtsorganismus auszuharren. „Dieses grundverschiedene Wachstumsverhalten hat bedeutenden Anteil am Krankheitsverlauf und entscheidet über den Erfolg von Antibiotikabehandlungen“, sagt Jörg Vogel.
Einzelne infizierte Makrophagen analysiert
Jedoch sei bis heute weitgehend unklar, wie einzelne Makrophagen auf die verschiedenen Lebensweisen der Salmonellen reagieren. Um hier Licht ins Dunkel zu bringen, hat das Würzburger Forschungsteam Makrophagen in Zellkultur gehalten und sie dann mit Salmonellen infiziert.
Danach wurden aus einzelnen Wirtszellen die RNA-Moleküle isoliert und mit Hochdurchsatzsequenzierung analysiert. Dabei ließen sich über 5.000 unterschiedliche RNA-Moleküle pro infizierter Wirtszelle nachweisen. Anschließend wurde diese Information über die Genexpression jeder einzelnen Zelle mit dem Wachstumsverhalten der Bakterien in Beziehung gesetzt.
Die Ergebnisse: Makrophagen, in denen einzelne, ruhende Bakterien sitzen, zeigen eine starke entzündungsfördernde Immunantwort. Sie schütten Botenstoffe aus, die wieder andere Immunzellen an den Ort der Infektion locken sollen. Damit reagieren sie nicht anders als Makrophagen, die nur äußerlich mit den Bakterien in Kontakt kommen. Das heißt: „Diese Makrophagen können die Bakterien in ihrem Inneren nicht erkennen, die fliegen dort sozusagen unter dem Radar“, erklärt Emmanuel Saliba, Erstautor der Studie.
Dagegen zeigen Makrophagen, die mit vielen, sich stark vermehrenden Salmonellen infiziert sind, eine komplett andere, nämlich eine anti-entzündliche Immunantwort. Ist das ein Trick der Salmonellen? Bringen sie die Makrophagen dazu, keinen Alarm zu schlagen, und halten sie sich damit die Immunabwehr vom Leib? Funktioniert dieser Trick vielleicht nicht immer? Gibt es dann doch Immunalarm, und die eingedrungenen Bakterien schalten notgedrungen auf ihr Ruheprogramm um?
Interessant für viele Gebiete der Biomedizin
„Was hier die Ursache und was die Wirkung ist, können wir leider noch nicht sagen, denn wir haben die Situation bislang nur zu einem einzigen Zeitpunkt nach der Infektion betrachtet“, erklärt Alexander Westermann, der ebenfalls in Vogels Team forscht. Weitere Studien seien nötig. Trotzdem gewähren diese ersten Ergebnisse schon jetzt neue Einblicke in die Wirtsantwort auf Krankheitserreger. Und mit der neuen Technik lassen sich bakterielle Infektionen in bis dato ungekannter Auflösung studieren – nämlich auf der Ebene einzelner Zellen.
Die in der Würzburger Core Unit Systemmedizin etablierte Single-cell-RNA-seq-Methode dürfte auch für viele andere Projekte der biomedizinischen Forschung von Interesse sein. „Damit lässt sich zum Beispiel auch die Heterogenität von Tumorzellen oder die Wirkung von Medikamenten in einzelnen Zellen mit bisher unerreichter Genauigkeit beschreiben“, so Professor Vogel.