Weltweit größtes Bakteriengenom sequenziert
Nur etwa ein Prozent des mikrobiellen Lebens um uns herum ist bisher im Labor kultiviert und beschrieben worden. Damit sind viele Potentiale der Mikroben noch unbekannt und nicht genutzt. Bestens untersucht waren lange Zeit nur Krankheitserreger wie multiresistente Staphylokokken oder biotechnologisch interessante E. coli-Stämme, die aber genetisch keineswegs repräsentativ sind. Forscher des Leibniz-Instituts DSMZ–Deutsche Sammlung von Mikroorganismen und Zellkulturen GmbH aus Braunschweig und des Joint Genome Institute (JGI) in Kalifornien erarbeiten deshalb seit drei Jahren systematisch die sogenannte „Genomic Encyclopedia of Bacteria and Archaea“ (GEBA), die wesentlich breiter aufgestellt ist. Im Zuge dieser Forschungsarbeiten wurde nun das Genom des Bodenbakteriums Ktedonobacter racemifer entziffert, welches weltweit das bisher größte beschriebene Bakteriengenom ist. Die Ergebnisse der Forscher sind in der letzten Ausgabe der Standards in Genomic Sciences veröffentlicht.
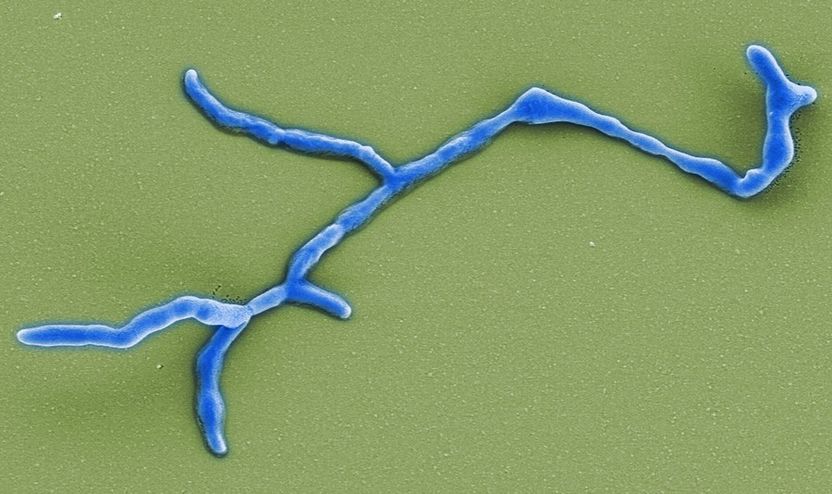
Elekronenmikroskopische Aufnahme des myzelbildenden Bodenbakteriums Ktedonobacter racemifer.
Manfred Rohde, Helmholtz-Zentrum für Infektionsforschung GmbH
„Im GEBA-Projekt untersuchen wir vor allem Mikroorganismen, die an „dunklen Flecken“ im Stammbaum der Bakterien stehen. Über die Auswertung von Genomdaten sind Rückschlüsse auf Verwandtschaftsverhältnisse zwischen Bakterien möglich“, erklärt PD Dr. Markus Göker, Bioinformatiker an der DSMZ. „Das Bakterium Ktedonobacter racemifer gehört zu den zehn, in Bezug auf ihre Stellung, wichtigsten Organismen im Bakterienstammbaum“.
„Das Genom des Gram-positiven Bodenbakteriums Ktedonobacter ist mit 11,453 Protein-codierenden Genen und 87 RNA-Genen das bisher größte dokumentierte Bakteriengenom“, erklärt Dr. Birte Abt, Wissenschaftlerin in der DSMZ-Abteilung Mikrobiologie. „Große Genome sind bei Bakterien mit einem
komplizierten Lebenszyklus mit Myzel- und Sporenbildung nicht ungewöhnlich. Dieses enthält aber auch sehr viele Transposons oder auch Wiederholungssequenzen, sogenannte „springende Gene“. Diese kleinen DNA-Stücke können ihre Position im Genom verändern. Ihre Funktion ist aber noch nicht vollständig geklärt.“
„Der Bakterienstamm wurde 2006 von einem Forscherteam in Italien isoliert und in der DSMZ in der öffentlichen Sammlung hinterlegt. Daher konnte der Organismus hier vermehrt und qualitative hochwertige genomische DNA für die Sequenzierung aufbereitet werden“, berichtet Dr. Birte Abt weiter. „Für uns war es wichtig, diese sehr isoliert im Stammbaum stehende Bakteriengruppe zu sequenzieren, die bisher noch sehr wenige Organismen umfasst. Damit besteht immer eine Möglichkeit, neue interessante Proteinfamilien mit unbekannter Funktion zu entdecken“, so Markus Göker. „Vollständige Genomdaten sind auch aussagekräftiger für die Klassifikation von Mikroorganismen, als die bislang meist praktizierte evolutionäre Einordnung der Bakterien über kleine molekulare Markerstücke, wie vor allem die ribosomale 16S RNA.“
Neben wertvollen Einsichten in die Evolution bakterieller Genomgrößen einerseits und komplexer Lebenszyklen andererseits, hilft die Sequenzierung dieses Rekordgenoms auch dabei, die stammesverwandtschaftlichen Beziehungen von Mikroorganismen zu klären und damit ihre taxonomische Einteilung zu verbessern. Sowohl Ktedonobacter-Kulturen als auch annotierte Genomdaten stehen nun für weitergehende Forschungen frei zur Verfügung.
Originalveröffentlichung
Non-contiguous finished genome sequence and contextual data oft he filamentous soil bacterium Ktedonobacter racemifer type strain (SOSP1-21T). Standards in Genomic Sciences 5:97-111, 2011.
Originalveröffentlichung
Non-contiguous finished genome sequence and contextual data oft he filamentous soil bacterium Ktedonobacter racemifer type strain (SOSP1-21T). Standards in Genomic Sciences 5:97-111, 2011.
Organisationen
Weitere News aus dem Ressort Wissenschaft

Holen Sie sich die Analytik- und Labortechnik-Branche in Ihren Posteingang
Ab sofort nichts mehr verpassen: Unser Newsletter für Analytik und Labortechnik bringt Sie jeden Dienstag auf den neuesten Stand. Aktuelle Branchen-News, Produkt-Highlights und Innovationen - kompakt und verständlich in Ihrem Posteingang. Von uns recherchiert, damit Sie es nicht tun müssen.