Neue Möglichkeiten für den Nachweis einzelner Moleküle
DNA-Origami-Faltung bildet einen intelligenten Verschluss für festkörperbasierte Nanoporen
Eine neu entwickelte Variante von festkörperbasierten Nanosensoren wurde mit ein paar Tricks aus der Bionanotechnologie verbessert, so dass die Möglichkeiten zur Messung von Einzelmolekülen und damit ein markierungsfreies Screening von Proteinen erweitert wurden. Forschern an der TU München ist es gelungen, die Funktionalität von Festkörper-Nanoporen zu verbessern, indem sie diese mit Nanoplättchen-Deckeln aus DNA als eine Art Verschluss versehen haben. Die Ergebnisse dieser Forschungsarbeiten wurden in der Zeitschrift Angewandte Chemie, International Edition, veröffentlicht.

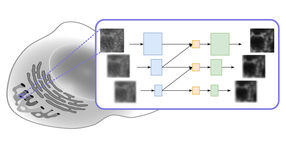
Diese Abbildung zeigt, wie ein DNA-Origami-Nanoplättchen als "Torwächter" für einen Festkörper-Nanopore-Sensor dienen kann.
TU München, Hendrik Dietz and Ulrich Rant
Zentrale Öffnungen in diesen Plättchen fungieren als „Torwächter“ und kontrollieren quasi einzelne Moleküle beim Passieren der Nanoschleuse. Gebildet werden diese Deckel mit dem sogenannten DNA-Origami-Verfahren: Abgeleitet von der japanischen Faltkunst Origami werden DNA-Stränge so synthetisiert, dass sie sich zu maßgeschneiderten Strukturen mit spezifischen chemischen Eigenschaften falten.
Im Verlauf der letzten Jahre ist es der Forschungsgruppe um Prof. Hendrik Dietz an der TUM gelungen, die DNA-Origami-Technik deutlich zu verfeinern. Dabei konnten sie zeigen, dass die mit Origami-Technik hergestellten Strukturen für Forschungszwecke in unterschiedlichsten Bereichen eingesetzt werden können. Ähnliche Fragen zur Messung einzelner Moleküle hat zur gleichen Zeit das Team von Dr. Ulrich Rant mit festkörperbasierten Nanoporen-Sensoren untersucht. Das messtechnische Prinzip dieser Sensoren basiert auf einer dünnen Halbleitermembran mit wenige Nanometer großen Öffnungen, durch die die gewünschten Biomoleküle einzeln geschleust werden. Wenn Biomoleküle durch diese Poren schlüpfen oder dort verweilen, liefern kleinste Änderungen des elektrischen Stroms, der durch die Nanopore fließt, Informationen über ihre charakteristischen physikalischen Eigenschaften. Inzwischen untersuchen Dietz und Rant gemeinsam – beide sind Fellows des TUM Institute for Advanced Study – welche Möglichkeiten eine kombinierte Anwendung dieser beiden Technologien eröffnen könnte.
Für das neue Messverfahren – das vor diesen Experimenten rein hypothetischer Natur war – wird zunächst ein DNA-Origami-Nanoplättchen über dem schmalen Ende einer konisch zulaufenden Festkörper-Nanopore positioniert. Durch eine Modifikation der Größe der zentralen Öffnung in der DNA-Nanoplatte lässt sich eine Filterung von Molekülen nach ihrer Größe erreichen. Um das System weiter zu verfeinern, werden Einzelstrang-DNA-Rezeptoren in der Öffnung als eine Art „Köder“ platziert, die sequenzspezifisch Zielmoleküle binden und damit den Nachweis einzelner Moleküle ermöglichen. Weitere denkbare Anwendungen sind hier unter anderem biomolekulare Interaktions-Screens und der Nachweis einzelner DNA-Sequenzen. Im Prinzip könnte man dieses System auch als Grundlage für ein neues Verfahren zur DNA-Sequenzierung nutzen.
Die Wissenschaftler untersuchten jede dieser Ideen Schritt für Schritt. Dabei konnten sie sowohl die Selbstorganisation von maßgeschneiderten DNA-Origami-Nanoplättchen als auch die anschließende elektrisch geleitete Positionierung über den Festkörper-Nanoporen belegen. Sie konnten auch zeigen, dass die größenabhängige Filterung von Biomolekülen und der Nachweis einzelner Zielmoleküle über spezifische „Köder“ funktionieren. „Wir freuen uns besonders darüber, dass wir mit unserem Köder aus spezifischen DNA-Sequenzen einzelne Moleküle herausfiltern und nachweisen konnten“, erläutert Dietz. „Denn neben DNA könnten sich auch eine Menge andere chemische Bestandteile des Stoffgemischs an der entsprechenden Stelle des DNA-Nanoplättchen unspezifisch anheften.“
Für den Einsatz bei hochspezifischen Messverfahren wie der DNA-Sequenzierung gilt es noch einige Hürden zu überwinden, erläutert Rant. „In zukünftigen Arbeiten müssen noch grundlegende Fragen geklärt werden, zum Beispiel in wie weit der direkte Transport von Ionen über die Origami Nanoplättchen die erreichbare Messgenauigkeit beeinflusst oder wie eine noch stabilere Verankerung der Nanoplättchen auf den Festkörperporen erzielt werden kann.“